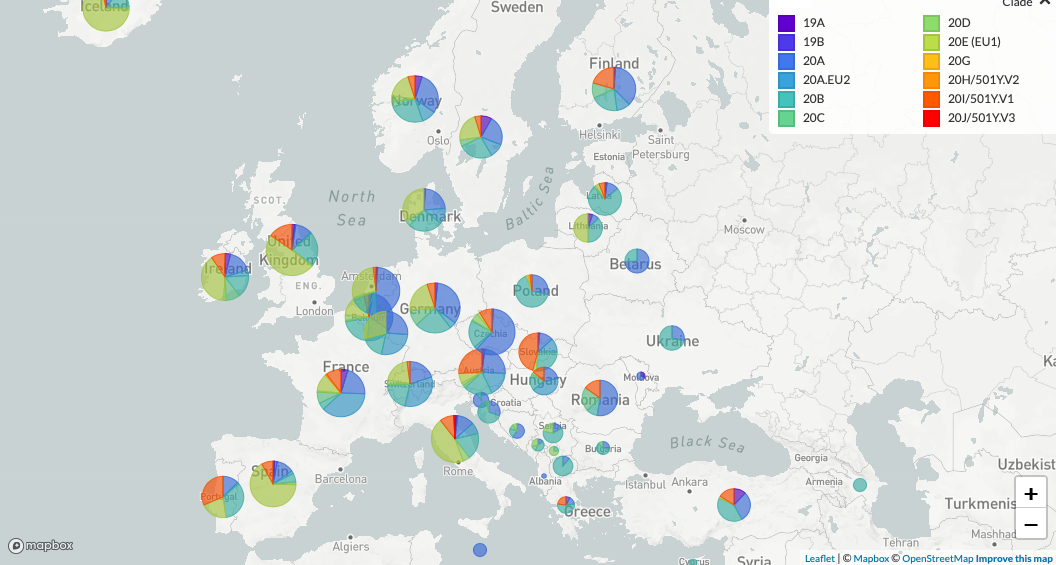
Los preprints son informes de investigación preliminares que no han sido evaluados por revisión por pares. Por este motivo, es preciso analizar bien su contenido y contrastar bien la información antes de confiar en ellos para guiar la práctica o informar en los medios de comunicación.
Realizada la lectura del preprint “Phylodynamics of SARS-CoV-2 transmission in Spain”, hacemos el siguiente resumen:
- El objetivo es analizar la difusión del SARS-Cov-2 en España y Europa.
- Los investigadores realizan el estudio filogenético mediante análisis bayesiano de las 28 primeras secuencias completas del genoma del virus en España y se comparan con otras 1590 secuencias completas del genoma del virus procedentes de Europa (Francia, Alemania, Italia y Finlandia) y Wuhan (China). Obtienen como resultado una mayoría de secuencias pertenecientes a los clusters G (13 secuencias) y S (13 secuencias) y, únicamente 2 secuencias con ramificación del cluster V. Estiman la presencia del los clusters S y G en nuestro país a mediados del mes de febrero.
- Los autores concluyen que en España existieron múltiples introducciones del SARS-CoV-2 y que al menos dos de ellas dieron como resultado la aparición de grupos de transmisión local, poniendo de manifiesto el extraordinario potencial del virus para producir una rápida y generalizada difusión geográfica.
- Los autores reconocen limitaciones del estudio que están relacionadas con la diversidad de estrategias y pasos para la secuenciación del genoma; con las diferentes técnicas de preparación de muestras utilizadas, con el enfoque metagenómico que implica un elevado número de lecturas de secuenciación y la informática empleada para analizar los datos. Todas estas razones podrían afectar a la calidad (los autores no pueden descartar la posibilidad de que se pierdan algunas moléculas de ARN) e introducir sesgos en la secuenciación y análisis, provocando diferencias genéticas entre muestras que sean debidas a la tasa de error y enmascarando otras diferencias genéticas reales.
Teniendo en cuenta todo lo anterior creo que, a día de hoy, no publicaría una noticia con esta información por varios motivos:
- Considero que las muestras son muy pequeñas para sacar conclusiones (solo 28 secuencias de España y 1590 con las cuales comparar). Según el proyecto Seq-COVID existen ya 6684 muestras secuenciadas y 14427 recibidas solamente de España.
- Ha pasado mucho tiempo desde la publicación del preprint y ha perdido relevancia. Como indico más adelante, algunos medios de información se hicieron eco de investigaciones similares al inicio de la pandemia y a fecha de hoy creo que hay cuestiones referentes al virus de mayor importancia.
- Si hacemos una búsqueda del autor principal en Google Scholar, encontramos que existe una versión nueva, revisada y publicada del estudio: A Founder Effect Led Early SARS-CoV-2 Transmission in Spain. Esta versión publicada solventa el problema anterior del tamaño muestral al incluir 12,511 secuencias de genoma completo obtenidas mundialmente e incluyendo 290 de España, procedentes de 11 localizaciones distintas. Si tuviese que elaborar una noticia sería a partir de este y no del preprint y me pondría en contacto con el grupo de investigadores para poder ofrecer mejor la información de un tema tan complicado como es la epidemiología genómica y tan sensible para una población que ya está bastante agotada por la situación y tanta información.
Si me encontrase en la situación de tener que tomar la decisión en abril, momento en el que se publicó el preprint, tendría dudas en cuanto a cómo proceder, pero creo que tampoco lo haría noticia:
- Por un lado, en esas fechas la información de la cual disponíamos era muchísimo menor, por lo que aumentaría el nivel de relevancia de estos hechos, haciendo el preprint algo más noticiable. Sin embargo, estando en pleno confinamiento y con una lluvia de datos y noticias constante, la única relevancia para un público general y no especializado es la rápida transmisión del virus, de lo cual ya teníamos noticias al respecto. La Agencia SINC se hacía eco el 25 de abril de otro proyecto español que también analizaba cómo y cuándo llegó el virus a nuestro país. Por aquel entonces, analizaba el genoma de 4000 muestras de 40 hospitales españoles y pretendía llegar a las 20000.
- Por otro lado, el problema de la muestra seguiría presente, por lo que, de publicar una noticia, haría referencia a esta cuestión y me pondría en contacto con alguno de los investigadores, tanto del grupo que publicó el preprint como del grupo de la noticia publicada en SINC, para comparar datos y entender mejor la información y las implicaciones de ambos estudios.
REFERENCIAS:
Díez-Fuertes, F., Iglesias-Caballero, M., Monzón, S., Jiménez, P., Varona, S., Cuesta, I., Zaballos, Á., Thomson, M. M., Jiménez, M., García Pérez, J., Pozo, F., Pérez-Olmeda, M., Alcamí, J., & Casas, I. (20 de abril, 2020). Phylodynamics of SARS-CoV-2 transmission in Spain. BiorXiv, 1-15. https://doi.org/10.1101/2020.04.20.050039. Preprint
Díez-Fuertes, F., Iglesias-Caballero, M., García-Pérez, J., Monzón, S., Jiménez, P., Varona, S., Cuesta, I., Zaballos, Á., Jiménez, M., Checa, L., Pozo, F., Pérez-Olmeda, M., Thomson, M. M., Alcamí, J., & Casas, I. (2020). A Founder Effect Led Early SARS-CoV-2 Transmission in Spain. Journal of Virology, 95(3), 1-15. https://doi.org/10.1128/jvi.01583-20
Hernando, A. (2020, 25 abril). “Queremos entender cómo se ha transmitido el coronavirus y estar preparados para futuros rebrotes”. Agencia SINC. https://www.agenciasinc.es/Entrevistas/Queremos-entender-como-se-ha-transmitido-el-coronavirus-y-estar-preparados-para-futuros-rebrotes
SeqCOVID – Genomic epidemiology of SARS-CoV-2 in Spain. (2021). Seq-COVID. http://seqcovid.csic.es
Imagen: Genomic epidemiology of novel coronavirus – Europe-focused subsampling on Nextstrain.